使用 Mol*¶
结构查看器¶
位于界面中央的结构查看器是 Mol* 的主要组件,负责显示当前打开的 PDB 文件中分子的 3D 结构。
鼠标控制¶
- 旋转:点击鼠标左键并移动。或者,使用 Shift 键 + 鼠标左键并拖动以旋转画布。
- 平移:点击鼠标右键并移动。或者,使用 Ctrl 键 + 鼠标左键并移动。在触摸屏设备上,使用两个手指拖动。
- 缩放:使用鼠标滚轮。在触摸板/触摸屏设备上,使用两个手指拖动。
- 居中和缩放:使用鼠标右键单击要聚焦的结构的部分。
- 裁剪:使用 Shift 键 + 鼠标滚轮来更改裁剪平面。在触摸板上,使用 Shift 键 + 两个手指拖动。
- 高亮:将鼠标悬停在 3D 画布中显示的 3D 结构的任何部分上(或悬停),而不点击它,将根据当前的选择粒度将其高亮显示(通过将其着色为品红色)。此外,在 3D 画布的右下角,为结构的高亮部分列出了有关 PDB ID、模型编号、实例、链 ID、残基编号和链名称的信息。
- 聚焦:在默认模式下,单击 3D 对象以聚焦它。聚焦的对象及其周围的对象将以球棍模型显示。所有局部非共价相互作用都会被标出。要隐藏周围的对象,只聚焦目标,请再次单击目标。要取消聚焦,请单击 3D 画布中任何没有原子的点。
按钮菜单¶
此菜单为用户提供了快速访问 3D 画布的一些常用操作。它位于 3D 画布的右侧,具有以下功能。
| 功能 | 描述 | 图标 |
|---|---|---|
| 重置视角 | 将 3D 画布上的结构居中并重置视角 | |
| 截屏 | 对当前视图截屏并提供分辨率和下载选项 | |
| 显示/隐藏控制面板 | 隐藏/显示右侧的控制面板 | |
| 全屏 | 将 3D 画布和控制面板扩展到完整的浏览器屏幕 | |
| 设置/控制信息 | 设置 3D 画布的视图,查看在 3D 画布中鼠标控制的帮助信息 | |
| 选择模式 | 在默认模式和选择模式之间切换 |
选择模式¶
点击按钮菜单中的""按钮即可切换到选择模式。
在选择模式下,单击残基(或 3D 中的任何对象)将选择它。将选择什么取决于选择粒度的值。结构的选定部分将在 3D 画布和序列查看器中以明亮的绿色显示。单击 3D 画布中任何没有原子的点将清空选区。
当您将选择粒度设为“Residue(氨基酸)”时,按住 Shift 键并依次点选起止残基可以沿聚合物序列选择片段。您也可以在序列查看器中按住鼠标、沿序列拖动再释放鼠标以选择多个残基构成的片段。
激活选择模式会在结构查看器的顶部打开一个工具栏。以下是工具栏的示例以及对每个按钮的描述:
| 功能 | 描述 | 图标 |
|---|---|---|
| (选择和高亮的)选择粒度 | 控制选择或高亮交互应用的级别(即原子、残基、链等) | |
| 选择并集 | 选择一个集合并将它添加到当前选区中 | |
| 选择差集 | 选择一个集合并将它从当前选区中减去 | |
| 选择交集 | 选择一个集合并取它和当前选区的交集 | |
| 重新选择 | 选择一个集合并设为当前选区 | |
| 应用主题到选区 | 允许将颜色、透明度和裁剪的更改应用到当前选区 | |
| 从当前选区创建组件 | 允许创建组件并独立于其他组件更改其表示 | |
| 从所有组件中移除选区 | 从 3D 画布中移除当前选区 | |
| 撤消修改选区 | 在保持选择模式打开的同时撤消某些操作(更改颜色、隐藏组件等) | |
| 显示/隐藏帮助 | 提供有关选择操作、表示操作和鼠标控件的摘要 | |
| 关闭选择模式 | 从选择模式切换到默认模式 |
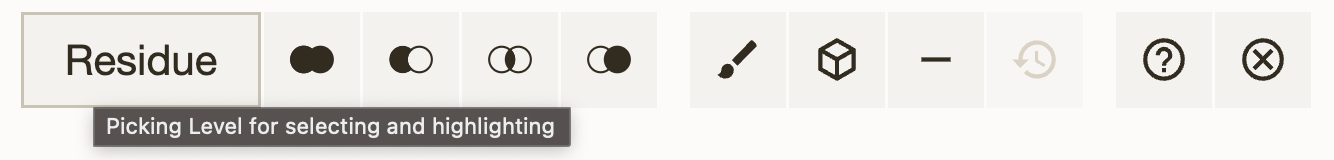
选择粒度¶
选择粒度决定了选择和高亮的粒度,或者说,每次选择和高亮操作是作用于一个原子、一个氨基酸(默认行为)、一条链、一个实体(entity)、一个模型(model)还是一个结构(structure)。例如,如果选择粒度设为 "Chain",当鼠标点击 3D 画布中的一个原子时,该原子所在的整条链将会被选中。如果结构中包含对称实例(symmetry instances),且选择粒度以 "instances" 结尾(例如 "chain instances"),操作将会包含选中原子 / 氨基酸 / 链的所有对称实例。
另外,选择粒度还会决定了序列查看器中的选择粒度,以及默认模式下聚焦操作的粒度。
选择粒度可以在选择模式工具栏(位于 3D 画布上方)中修改。
从选区创建组件¶
在选择模式下,我们可以从选区创建新组件。创建组件让用户能对结构的某些部分进行单独操作(详情请看操作组件)。例如,如果您想单独可视化蛋白质和配体,您可以创建一个“配体”组件并隐藏它。然后,您可以单独更改蛋白质的表示,而不必担心配体的干扰。
您可以单击位于结构查看器顶部、选择模式工具栏中的""按钮来创建组件。点击后,您将看到一个对话框,您可以在其中设置如下参数:
- Selection(选区):用于创建组件的选区,默认为当前选区。您也可以使用其他选区,例如所有氨基酸构成的选区(以ALA为例,您可以单击"Amino Acid > Alanine (ALA)"),或者当前选区周围 5 Å 内的所有残基(您可以单击"Manipulate Selection > Surrounding Residues (5Å) of Selection")。
- Representation(表示):在创建组件的同时为其添加的结构表示。默认为空(无表示)。
- Options(选项):
- Label(标签):组件的标签。默认为“Label”。
- Check existing(查重):如果启用,Mol* 将检查是否已存在具有相同内容的组件,以避免您创建重复的组件。默认不开启。
案例:从复合物结构中提取抗体链并保存¶
如果您想要提取一个抗体-抗原复合物结构中的抗体链并将其保存到另一个文件中,您可以按照以下步骤操作:
1. 从云端导入或者从本地上传复合物结构文件。在文件与任务管理器面板中点击文件名以打开该文件。
2. 点击结构查看器右侧的""按钮,进入选择模式。
3. 将选择粒度设为"Chain",然后点击需要保存的抗体链。
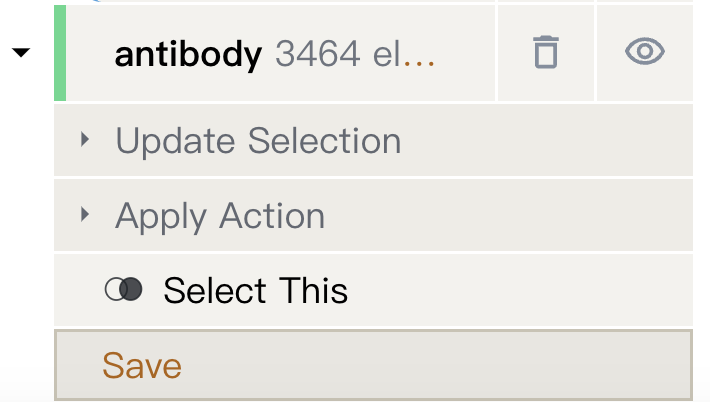
4. 点击结构查看器顶部的"
"按钮,创建一个名为"antibody"的组件。
5. 点击结构对象面板中的"antibody"组件,然后在下拉菜单中点击"Save",将其保存为项目中的一个 CIF 文件。输入文件名并点击"√"。
{ style="width:250px; margin: 0 auto;" }
- 现在,在文件与任务管理器面板中将会出现一个新的 CIF 文件。
序列查看器¶
序列查看器显示上传到 Mol* 中的 PDB 结构中存在的大分子(蛋白质和核酸)的聚合物序列。此外,它还提供了快速访问结构中存在的任何小分子配体或实体的方法。它位于 3D 画布上方的屏幕顶部。
序列查看器包含几个下拉菜单,允许用户选择要显示的序列:
- 结构 (structure):选择要显示的结构。
- 模式 (mode):在链(chain)、聚合物(polymers)和全部(everything)三种显示模式之间选择。在链模式中,每个链都单独显示。在聚合物模式中,所有聚合物都一起显示。在全部模式中,所有实体都一起显示。
- 实体 (entity, 仅在链模式中出现):选择要显示的实体。
- 链 (chain, 仅在链模式中出现):选择要显示的链。

序列查看器的右上方有四个按钮:
(复制到剪贴板):将显示的序列复制到剪贴板。目前,仅在显示模式设置为“chain”(链)时才显示此按钮。
(序列自动换行):将序列换行以适应面板的宽度。如果禁用,序列将显示在一行中。默认启用。
(显示二级结构):显示序列的二级结构注释。序列中的α-螺旋将以波浪线标出,β-折叠以直下划线标出。默认禁用。
(显示 CDR 注释):显示序列的 CDR(互补决定区)注释。默认禁用。单击后,将弹出一个菜单供您选择要使用的 CDR 定义。支持的 CDR 定义包括“IMGT”、“Chothia”、“Kabat”和“North”。当您确认后,相应的 CDR 残基将在序列中用下划线标出。
结构对象¶
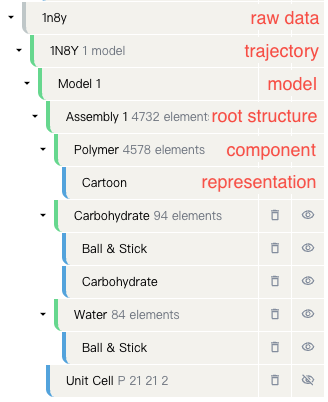
结构对象是在结构查看器中显示的3D对象。您可以通过位于项目编辑器左下角的结构对象面板来访问它们。结构对象组件包含了当前已打开的文件列表(在文件与任务管理器中具有淡蓝色背景的文件)。 每个文件都以树状结构显示。他们具有以下层次结构:
结构的层级划分¶
-
原始数据:文件的原始数据,通常为 PDB 或 mmCIF。如果文件是从云端导入的,此信息可能缺失。
-
轨迹:由一组模型构成。它表示结构按时间顺序变化的轨迹,或者表示一系列相关结构的集合。这是结构层级划分的顶层。
-
模型:轨迹中的一个帧,它包含某些分子的结构信息。您可以通过点击“轨迹 -> 更新分子模型”并选择您想要显示的模型。
-
结构:一个结构是一系列单元信息的集合。通常情况下,一个单元对应一条链。除了连续的具有相同 entity_id 和相同 auth_asym_id 的“单原子链”,例如水分子。
构建根结构¶
根结构是由一系列组件或子结构构成的层级结构。 当您在 mmCIF/PDB 文件中进行适当注释后,您便可以通过多种方式从模型构建根结构。
-
模型(Model):仅基于模型中的原子坐标和化学键信息创建结构,不考虑任何更高级的晶体结构排列信息。
-
组装(Assembly):基于用户选择的大分子组装方式来创建结构。例如,在晶体结构中,我们通过将对称操作应用于非对称单元来创建晶胞的晶体学组装。
-
对称(Symmetry):通过将用户定义的对称操作(包括平移、旋转或镜像操作)应用于非对称单元来创建结构。
-
对称伴体(Symmetry Mates):通过在一定半径内找到对称伴体来创建结构。对称伴体是通过对称操作创建的非对称单位的镜像。
-
对称组装(Symmetry Assembly):使用定义的对称操作和非对称单元来创建自定义的生物学组装。
-
自动(Auto,默认行为):如果没有可用的组装,则创建一个“模型”结构。否则,默认展示第一个组装。
您可以通过点击“根结构 -> 更新结构”并选择所需的结构构建方法来更改构建根结构的方式。
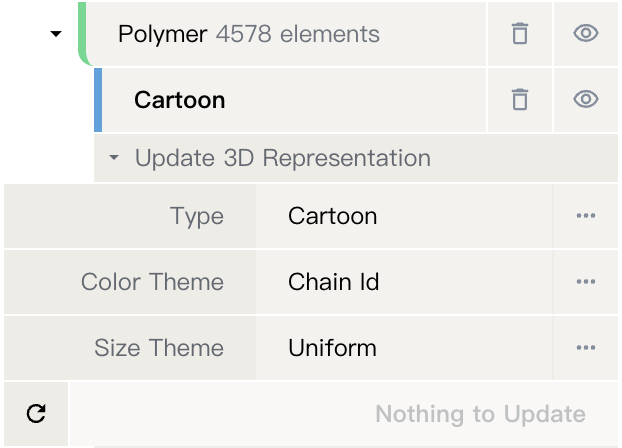
结构表示¶
结构的表示方式确定了结构在结构查看器中的显示方式。 以下是几种结构的表示方式:
-
Cartoon(卡通):以流畅的方式显示多聚物的迹线原子的带状、块状、管状结构。
-
Backbone(主链):以圆柱和球体显示多聚物的主链。
-
Ball & Stick(球棍):以球体显示原子,以圆柱显示键。
-
Carbohydrate(碳水化合物):以符号(3D SNFG)显示碳水化合物。
-
Ellipsoid(椭圆体):显示原子元素的各向异性位移椭圆体,以及以圆柱显示键。
-
Gaussian Surface(高斯表面):显示高斯分子表面。
-
Gaussian Volume(高斯体积):使用直接体积渲染显示高斯分子密度。
-
Labels(标签):为原子、残基和链显示标签。
-
Lines(线条):以线条显示键,以点或十字显示原子。
-
Molecular Surface(分子表面):显示分子表面。
-
Orientation(取向):为多聚物链显示取向椭球体。
-
Point(点状):将元素(原子、粗粒球体)显示为点。
-
Putty(软膏):以流畅的方式显示多聚物的迹线原子的管状结构。
-
Spacefill(空间填充):将原子/粗粒元素显示为球体。
-
Non-covalent Interactions(非共价相互作用):将非共价相互作用显示为虚线圆柱体。
-
Validation Clashes(验证冲突):将原子之间的冲突显示为圆盘。数据来自 wwPDB 验证报告,通过 RCSB PDB 获取。
-
Membrane Orientation(膜取向):使用 ANVIL 算法计算的膜取向。
除表示方式外,还有两个关键参数确定了结构的显示方式:颜色主题决定着色,大小主题决定表示的大小。
默认的结构表示¶
默认的结构表示方式决定了根结构如何被分成组件,以及每个组件的默认显示方式。 决定显示方式主要有两个步骤:
-
确定大小:结构的大小被分为五个类别:小、中、大、巨大和庞大。
-
具有少于 10/5000/30000 残基的结构分别被认为是“小”、“中”和“大”。
-
如果结构的残基数大于 30000,如果它有较高的对称单元数,它将被归类为“巨大”;否则,它将被被归类为“庞大”。
-
-
选择表示方式:根据确定的大小,选择适当的表示方式。逻辑如下:
-
小:使用原子细节(AtomicDetail)显示并显示碳水化合物符号。
-
中:如果残基和间隔比大于3,则使用聚合物与配体(PolymerAndLigand)显示。如果小于3,则以与“小”相同的方式显示。
-
大:使用聚合物卡通(PolymerCartoon)显示,其中配体不显示。
-
巨大/庞大:使用粗粒表面(CoarseSurface)显示,这有助于在不过载系统的情况下显示这种大小的结构。
-
此自动化过程可以根据分子结构的大小和复杂性提供最佳视图,以此来平衡展示的细节和性能。
操作组件¶
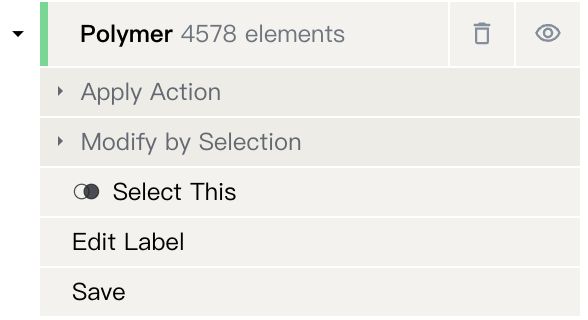
以下操作可在组件上执行:
-
显示/隐藏:通过“
/
”按钮显示/隐藏组件。
-
删除:通过“
”按钮删除组件。这将取消分组,但结构将保持完整。
-
更新表示:通过点击“应用操作 -> 更新3D表示”,然后选择所需的表示类型、颜色主题和大小主题更新表示。
-
修改:通过点击 “应用操作 -> 按选择修改”,然后选择以下操作之一来修改组件的内容:
-
包括:新组件将是当前组件与另一个组件的并集。
-
减去:新组件将是当前组件中除另一个组件中的部分之外的部分。
-
交叉:新组件将是当前组件与另一个组件的交集。
-
-
编辑标签:通过点击 “应用操作 -> 编辑标签”,然后在文本框中输入新标签来编辑组件的标签。
-
保存:通过点击 “应用操作 -> 保存”,然后在文本框中输入所需的文件名来将组件保存为项目中的文件。